Es wimmelt von Infos und fake news in allen Medien.
Hier finden sich veröffentlichte, aber irgendwie wenig sichtbare Informationen.
Nein, keine in der Art:
Verschwörer in der Wall Street oder im Mossad oder beim Bilderberg hätten ein Virus in die Welt gesetzt, um sich diese untertan zu machen.
Keine rassistischen Dummheiten wie die vom "chinesischen Virus".
Keine Behauptungen, wir hätten es gerade mit einem simplen Schnupfen zu tun.
Sondern solche, die helfen, einen kritischen Abstand zu regierungsamtlichen Verlautbarungen zu halten.
Denn erinnern wir uns: Es sind die gleichen Experten und Regierenden, die gestern unser Gesundheitssystem planmäßig (nicht etwa nur fahrlässig) ruiniert haben, die uns jetzt vorschreiben, was richtig und was verboten ist. Und Vorsicht: Die Grundhaltung ist links, auch wenn hier merkwürdige Positionen in der Linken befragt werden.
Übersetzungen aus dem Englischen sind oft holprig, weil mit dem Google Übersetzer (inzwischen deepl.com) vorgenommen.
Zitate aus dem Internet haben als Abrufdatum das des Beitrags.
Über den Button oben kann ein RSS-Feed abonniert werden.
Hier kann man die neuesten Beiträge via -Telegram erhalten.
Dieser Blog benötigt keine Spenden! Trotzdem vielen Dank für die Angebote.

WOW. Was für ein Erfolg 🙂 !!
https://twitter.com/jeha2019/status/1522323851031552003
https://twitter.com/LViehler/status/1522309894061404160
https://twitter.com/USMortality/status/1522300985422536704
Hallo Herr Aschmoneit,
können sie mir erklären, wieso die Weltbank Exporte von Testkits, die essenziell für den Kampf gegen COVID-19 seien (ehemals als COVID-19 Testkits gelabelt), bereits für 2017 anführt?
Für 2017 stehen sind dort deutsche Exporte mit einem Volumen von 17.5 Mrd USD angegeben.
Für 2020 23 Mrd USD
Für 2016 0 USD.
Wenn ich mich nicht irre, so müssen die Nukleotidsequenzen die getestet werden bei der Herstellung des Testkits bereits feststehen.
Was sind das also für Testkits die dort exportiert wurden?
Ich danke ihnen jetzt schonmal für ihre Hilfe.
https://wits.worldbank.org/trade/comtrade/en/country/ALL/year/2017/tradeflow/exports/partner/WLD/nomen/h5/product/300215
Hängt es leicht hiermit zusammen?
https://www.nature.com/articles/nm.4131.pdf
Follow-Up 2019:
https://gh.bmj.com/content/4/Suppl_2/e001105
@Impfadmiral: PCR ist ein Standardverfahren in der medizinischen Diagnostik und wurde schon vor Corona vielfach angewendet, vgl. https://de.wikipedia.org/wiki/PCR#Medizin_und_Diagnostik
Die Umbenennung einer 2017 eingeführten Welthandelsstatistik-Warengruppe Anfang 2020 aufgrund von Covid-19 und die unklare Abgrenzung bzw. Bezeichnung der Warengruppen 3822.00 und 3002.15 wurden im Sommer 2020 intensiv diskutiert, vgl. z. B. https://healthfeedback.org/claimreview/covid-19-test-kits-were-not-purchased-in-2017-and-2018-claim-is-based-on-mislabeled-data/
Ein kleiner Junge, der hieß Christian,
der fand es meistens ganz gemein,
wenn sich Erwachs'ne unterhielten
und sagten: Du bist noch zu klein!
Dann wählten sie ganz schwere Worte
und haben über ihn gelacht.
Doch eines Tages hat sich Christian,
auch was verzwicktes ausgedacht:
Pieksi-Taxi
Nuvovaxi,
Coronation.
Spikie Spoxi
Immunotoxi,
Perkulation!
…
Nach 30 Jahren dann im Fernsehn
da sah man einen klugen Mann.
Er hieß Prof. Dr. Drosten.
Den riefen viele Leute an.
Er hatte immer eine Antwort,
was ihn die Leute auch gefragt.
Er krault sich einmal seine Haare
und hat bedeutungsvoll gesagt:
Pieksi-Taxi …
Frei nach Gerhard Schöne:
https://www.youtube.com/watch?v=xKs5Vh-zdac
Sehr schön, aber ich glaube es heißt Perkolation. Muss fairer Weise gestehen, dass ich das Wort selbst nachschlagen musste. Sonst hätte ich es nicht bemerkt. 😉
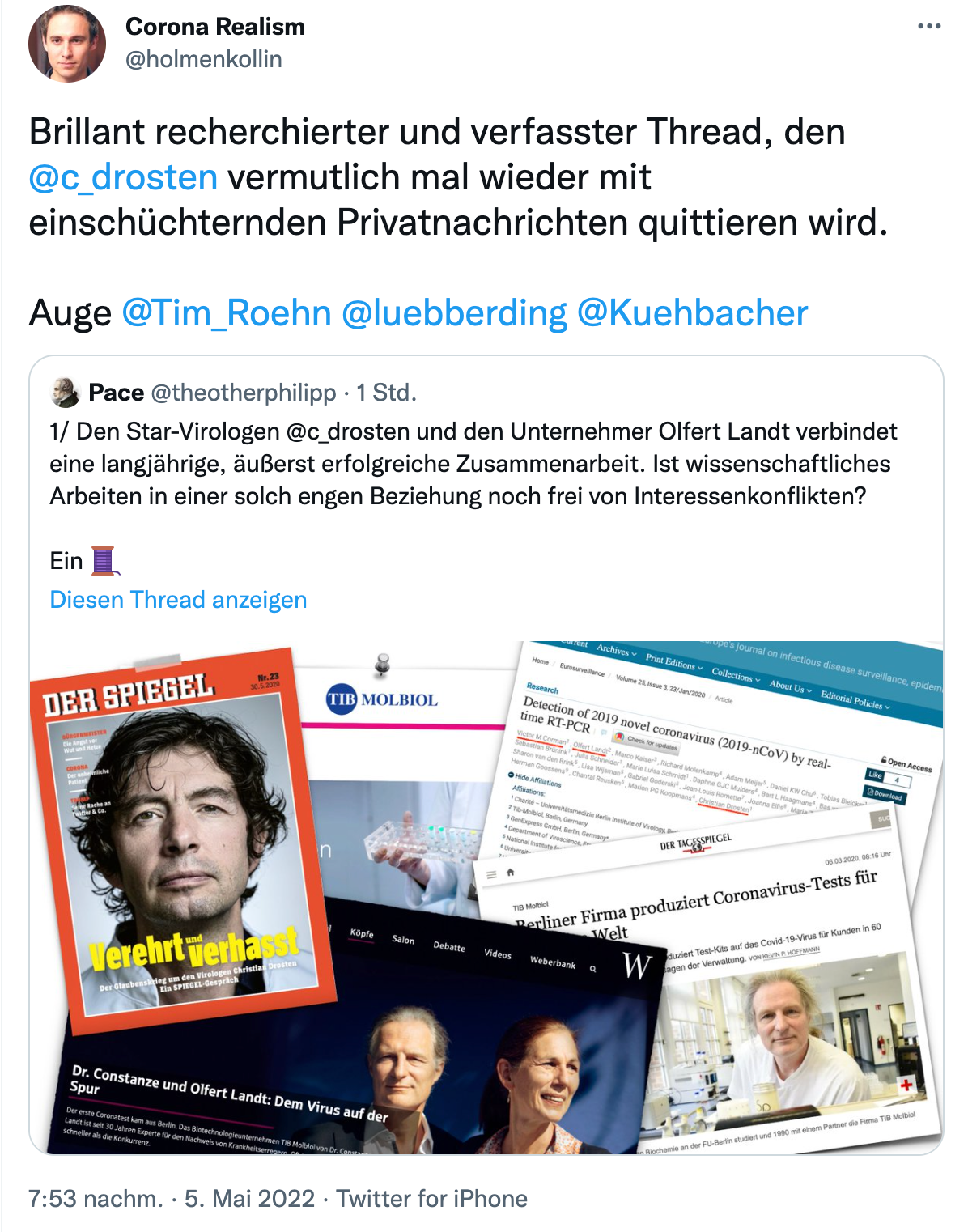
Zum Pace-Thread: https://threadreaderapp.com/thread/1522263132038733827.html
Dabei werfen die Ereignisse mehr Fragen auf, als sie beantworten. Ein Beispiel: Erst am 10.01. taucht weltweit die erste #SARSCoV2-Gensequenz bei virological.org auf. Woher hatte Landt das Wissen, die Reagenzien taggleich zu versenden?
Angeblich lief es so ab:
At 8am the following Saturday [Sydney, 2020-01-11, ungefähr 08:00 UTC+10 = 22 Uhr Vortag (10.01.) in UK, 23 Uhr in Berlin], sitting in his home study, Professor Holmes called Professor Zhang to ask a burning question: "Should we release this sequence?" He took a moment, and called his Australian colleague back. "He said: ‘OK, let's release it’," Professor Holmes said.
He then woke his colleague in Edinburgh, Professor Andrew Rambaut, who runs the website virological.org. "I told him we need to get this on online ... I was shaking as I pressed send, and off it went." It took 52 minutes from receiving the code from Professor Zhang to publishing – Professor Holmes timed it.
https://www.smh.com.au/national/nsw/virus-rebel-20201026-p568qj.html
Der maßgebliche Eintrag https://virological.org/t/novel-2019-coronavirus-genome/319 von Rambaut auf Virological hat den Timestamp 2020-01-11T01:05:33+00:00, gleich 01:05 Uhr am 11.01.2020 britische Zeit (UTC), 02:05 Uhr an der Charité und 11:05 Uhr in Sydney.
Wenn Rambaut seinem Post voranstellt:
10th January 2020
This posting is communicated by Edward C. Holmes, University of Sydney on behalf of the consortium led by Professor Yong-Zhen Zhang, Fudan University, Shanghai
dann hat er den Post wohl noch am 10.01. vor Mitternacht (UTC) vorbereitet, aber wohl erst um 01:05 online gestellt. (Mitternacht in Greenwich entspricht 10:00 Uhr in Sydney.) Also scheinbar doch etwas mehr als nur 52 Minuten von China über Australien nach England.
Das am 27.01.2020 editierte Posting verweist jedoch aktuell nur noch auf die NCBI-Datenbank, wo der Eintrag der ersten Version der Sequenz https://www.ncbi.nlm.nih.gov/nuccore/MN908947.1 mit 2020-01-12 datiert ist (Sonntag) und in den Angaben dazu steht:
AUTHORS Zhang,Y.-Z., Wu,F., Chen,Y.-M., Pei,Y.-Y., Xu,L., Wang,W., Zhao,S., Yu,B., Hu,Y., Tao,Z.-W., Song,Z.-G., Tian,J.-H., Zhang,Y.-L., Liu,Y., Zheng,J.-J., Dai,F.-H., Wang,Q.-M., She,J.-L. and Zhu,T.-Y.
TITLE Direct Submission
JOURNAL Submitted (05-JAN-2020) Department of Zoonoses, National Institute of Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Changping Liuzi 5, Beijing 102206, China
Der sequenzierte Virus lag Prof. Zhang und Team also wohl bereits am Sonntag, 05.01.2020, vor, an dem Tag hat Prof. Zhang angeblich auch gleich seinen Kollegen Edward Holmes in Sydney telefonisch über das Ergebnis der Sequenzierung informiert:
On Sunday, January 5, 2020, Professor Holmes was in a car headed for a Sunday brunch on Sydney's northern beaches when his phone pinged. Professor Zhang had received the virus sequence back from the lab and it looked a lot like SARS, the virus responsible for the 2003 outbreak in China. "I sort of swore down the phone — we thought it was like SARS back again." Others had also sequenced the new virus's genome, but Chinese authorities were being strict on what information could be released, Professor Holmes says.
https://www.abc.net.au/news/100552752
Da ist nur die Frage, warum China die Sequenz nicht selbst veröffentlicht, sondern auf diesem Wege weitergereicht hat, so dass Holmes und Rambaut neben Zhang auch zu Helden wurden, und was der Grund für die Verzögerung und die angeblich zwischen Zhang und Edwards getroffene Entscheidung über den (unvorbereiteten?) Veröffentlichungstermin erst am folgenden Samstagmorgen war.
Vermutlich drei Minuten nach Veröffentlichung auf Virological sandte Edward Holmes einen Tweet darüber in die Welt: https://twitter.com/edwardcholmes/status/1215802670176276482 dessen Timestamp im Screenshot unter https://www.asiapacific.ca/publication/who-knew-what-when-international-transmission-information geht vermutlich auf die US-Timezone UTC-5 zurück, wo es noch 10.01.2020 um 20:08 Uhr abends war.
Die WHO präsentierte am 21.01.2020 alternative Fakten:
• On 11 and 12 January 2020, WHO received further detailed information from the National Health Commission China that the outbreak is associated with exposures in one seafood market in Wuhan City.
• The Chinese authorities identified a new type of coronavirus, which was isolated on 7 January 2020.
• On 12 January 2020, China shared the genetic sequence of the novel coronavirus for countries to use in developing specific diagnostic kits.
https://www.who.int/docs/default-source/coronaviruse/situation-reports/20200121-sitrep-1-2019-ncov.pdf
Zurück nach Berlin.
Landt tippte auf ein Grippevirus, Christian Drosten auf Corona. Stündlich wurden die Datenbanken abgefragt. Als endlich die Gensequenz veröffentlicht wurde, schritten die Charité-Virologen und Landt zur Tat. Der Entwurf für einen neuen Test dauert nur ein, zwei Stunden. (...) setzte sich Landt an die Arbeit, konzipierte ein synthetisches Gen als Kontrolle – es gab in Europa ja noch kein Virus zum Ausprobieren – und gab alle Komponenten in die Synthese. Nach gut einer Nacht waren die Bausteine fertig. Was dann kam, war einerseits Routine (...)
https://weberbank-diskurs.de/dr-constanze-und-olfert-landt-dem-virus-auf-der-spur
Dem Tagesspiegel sagte Landt bekanntlich:
Die ersten [Tests] gingen bereits am 10. Januar per Luftpost nach Hongkong. Da war der Beipackzettel noch gar nicht fertig. „Den haben wir per Mail hinterhergeschickt“.
https://www.tagesspiegel.de/berlin/tib-molbiol-berliner-firma-produziert-coronavirus-tests-fuer-die-ganze-welt/25602142.html
Auch dieser Thread scheint ganz interessant in Bezug auf den zeitlichen Verlauf: https://flutrackers.com/forum/forum/-2019-ncov-new-coronavirus/china-2019-ncov/821830-
Auf Seite 11 bzw. in Posting 152 findet sich die Virological-Meldung mit Timestamp Jan 10, 2020, 21:50 GMT-5, also 02:50 morgens GMT/UTC.
Dort auch die URL der ursprünglich hinterlegten Sequenz: https://virological.org/uploads/short-url/34T6zCGO00f9RCFaeN9oe8g6r1p.gz
Thread https://virological.org/t/novel-2019-coronavirus-genome/320 ist auf virological.org nicht (mehr) verfügbar, der nächste Thread 321 wurde am 15.01.2020 von Marion Koopmans erstellt, im Team mit Bas Oude Munnink und anderen: „Initial assessment of the ability of published coronavirus primers sets to detect the Wuhan coronavirus“:
We have run a software algorithm that allows the alignment of large set of genomes against any primerset, to analyse a selected set of available PCR protocols for testing SARS-like viruses for mismatches with the new Wuhan coronavirus.
https://virological.org/t/initial-assessment-of-the-ability-of-published-coronavirus-primers-sets-to-detect-the-wuhan-coronavirus/321
Die verwendete Virus-Sequenz wird mit o.g. Virological-Veröffentlichung angegeben, außerdem 5 weitere GISAID-Sequenzen unter https://gisaid.org/CoV2020, die jedoch von Rambaut erst am Sonntag, 12.01.2020, um 12:14 auf Virological angekündigt wurden (in Thread 319). Abspann:
ErasmusMC, Rotterdam, The Netherlands:
Bas Oude Munnink, Richard Molenkamp, Reina Sikkema, Zsofia Igloi, Mart Lamers, Annemiek Baltissen – van der Eijk, Bart Haagmans, Ron Fouchier, Marion Koopmans
With the help of:
Hong Kong University, Hong Kong:
Daniel Chu, Malik Peiris
Im Anhang https://virological.org/uploads/short-url/v0PMNEiLpamGR0GObV2IQnaOU0P.zip finden sich die Dateien zu den Primern und Testergebnissen. Anhand einzelner Merkmale lässt sich nachweisen, dass die Software-Durchläufe bereits am Donnerstag, 09.01.2020, und Freitag, 10.01.2020, stattfanden, und nicht erst nach öffentlicher Bekanntgabe der Virus-Sequenzen am Samstag, 11.01.2020 (jeweils MEZ).
Vorgenannte doch nicht ganz wenige Personen hatten also scheinbar vorab von China Zugang zu deren Sequenzierungen erhalten.
Das erklärt auch, warum Victor Corman, Tobias Bleicker, Sebastian Brünink, Christian Drosten, Olfert Landt, Marion Koopmans, Maria Zambon, Malik Peiris bereits am Montag, 13.01.2020 ihr vorläufiges PCR-Protokoll veröffentlichen und darin schreiben konnten:
For oligonucleotide design and in-silico evaluation we downloaded all complete and partial (if >400 nucleotides) SARS-related virus sequences available at GenBank by January 1st, 2020. The list (n=729 entries) was manually checked and artificial sequences (lab-derived, synthetic etc.), as well as sequence duplicates removed, resulting in a final list of 375 sequences. These sequences were aligned and the alignment used for assay design. The alignment was later complemented by sequences released from the Wuhan cluster.
https://www.who.int/docs/default-source/coronaviruse/wuhan-virus-assay-v1.pdf
Obige Virological-Veröffentlichung vom 15.10. beschreibt also nachträglich einen Arbeitsschritt (Ausschluss übereinstimmender Primer) vor der Erstellung des PCR-Protokolls. Etwas kryptisch wird am Mittwoch, 15.10.2020, auf die bereits viel weitergehenden Erkenntnisse verwiesen:
Specific Wuhan coronavirus primer and probe sets have been developed in China and by Charité in Berlin. The primer set developed by Charité is currently tested by laboratories in Berlin, London, Hongkong and Rotterdam.
Der Rest ist Geschichte: https://www.eurosurveillance.org/content/10.2807/1560-7917.ES.2020.25.3.2000045 und komplementär z. B. https://www.thomaskubo.de/das-pcr-desaster.html oder https://www.cormandrostenreview.com/addendum/
Eine Zeitnovelle:
A novel coronavirus currently termed 2019-nCoV was officially announced as the causative agent by Chinese authorities on 7 January. A viral genome sequence was released for immediate public health support via the community online resource virological.org on 10 January (Wuhan-Hu-1, GenBank accession number MN908947 [2]), followed by four other genomes deposited on 12 January in the viral sequence database curated by the Global Initiative on Sharing All Influenza Data (GISAID).
https://www.eurosurveillance.org/deliver/fulltext/eurosurveillance/25/3/eurosurv-25-3-5.pdf
Zwischen Sonntag und Montag scheint dann an einigen Orten noch eine ganze Menge gelaufen zu sein:
Cell culture supernatants containing typed coronaviruses and other respiratory viruses were provided by Charité and University of Hong Kong research laboratories. Respiratory samples were obtained during 2019 from patients hospitalised at Charité medical centre and tested by the NxTAG respiratory pathogen panel (Luminex, S´Hertogenbosch, The Netherlands) or in cases of MERS-CoV by the MERS-CoV upE assay as published before [10]. Additional samples were selected from biobanks at the Rijksinstituut voor Volksgezondheid en Milieu (RIVM), Bilthoven, at Erasmus University Medical Center, Rotterdam, at Public Health England (PHE), London, and at the University of Hong Kong. Samples from all collections comprised sputum as well as nose and throat swabs with or without viral transport medium. Faecal samples containing bat-derived SARS-related CoV samples (identified by GenBank accession numbers) were tested: KC633203, Betacoronavirus BtCoV/Rhi_eur/BB98–98/BGR/2008; KC633204, Betacoronavirus BtCoV/Rhi_eur/BB98–92/BGR/2008; KC633201, Betacoronavirus BtCoV/Rhi_bla/BB98–22/BGR/2008; GU190221 Betacoronavirus Bat coronavirus BR98–19/BGR/2008; GU190222 Betacoronavirus Bat coronavirus BM98–01/BGR/2008; GU190223, Betacoronavirus Bat coronavirus BM98–13/BGR/2008. All synthetic RNA used in this study was photometrically quantified.RNA was extracted from clinical samples with the MagNA Pure 96 system (Roche, Penzberg, Germany) and from cell culture supernatants with the viral RNA mini kit (QIAGEN, Hilden, Germany).
https://www.eurosurveillance.org/deliver/fulltext/eurosurveillance/25/3/eurosurv-25-3-5.pdf
Refrain (zum Mitsingen und Mitschreiben):
Stündlich wurden die Datenbanken abgefragt. Als endlich die Gensequenz veröffentlicht wurde, schritten die Charité-Virologen und Landt zur Tat. Der Entwurf für einen neuen Test dauert nur ein, zwei Stunden.
https://weberbank-diskurs.de/dr-constanze-und-olfert-landt-dem-virus-auf-der-spur